摘要:
合集:AI案例-CV-医疗
AI问题:图像分割
数据集:细胞膜数据集
数据集价值:从显微镜图像中精确分割出细胞膜边界。
解决方案:U-Net模型、Tensorflow1.x开发框架。
一、问题和U-Net模型
图像语义分割是一项将数字图像中的每个像素都进行分类,并为每个像素分配一个类别标签的任务。其核心目标是让机器能够像人一样“理解”图像的构成。生物医学图像分割的任务目标是从显微镜图像中精确分割出细胞膜边界。应用于细胞生物学、医学研究、病理学分析。典型数据包括电子显微镜图像、荧光显微镜图像。
计算机视觉领域中的主要解决的问题和区别如下:
| 任务名称 | 输入 | 输出 | 目标 |
|---|---|---|---|
| 图像分类 | 一张图片 | 一个标签 | 图片里是什么?(整张图是“猫”) |
| 目标检测 | 一张图片 | 多个边界框和标签 | 物体在哪里?是什么?(图里有“猫”和“狗”) |
| 语义分割 | 一张图片 | 一张像素级标签图 | 每个像素属于什么?(区分出“猫”区域和“狗”区域) |
| 实例分割 | 一张图片 | 区分个体的像素级标签图 | 每个像素属于哪个个体?(区分出“猫1”、“猫2”和“狗”) |
1、U-Net模型
U-Net模型由Olaf Ronneberger等人于2015年在论文《U-Net: Convolutional Networks for Biomedical Image Segmentation》中提出。其最初的目标是解决在医学影像中精确分割细胞、组织等目标物的难题。这类任务通常需要高精度的像素级定位。其核心思想是设计一个能够同时捕获上下文信息(知道目标是什么)和位置信息(知道目标在哪里)的对称编码器-解码器结构。U-Net 但因其卓越的性能和简洁的架构,迅速成为整个计算机视觉领域,尤其是语义分割任务的基石模型。
U-Net的结构因其形状酷似英文字母“U”而得名,它由两条路径组成:
- 下采样路径 ,位于模型的左侧,为编码器。其作用是逐步提取图像特征,捕获上下文信息。随着网络层数的加深,特征图的分辨率降低,但通道数K增加。通道数K直接对应于你希望模型识别出的物体类别的数量。学到的特征从简单(如边缘、纹理)变得复杂抽象(如器官、结构)。其技术实现是由重复的两个3×3卷积(不填充)+ ReLU激活函数 和一个2×2最大池化(用于下采样) 块组成。
- 上采样路径,位于模型的右侧,为解码器。其作用是逐步恢复特征图的空间分辨率和细节信息,实现精确定位。其技术实现是由重复的上采样(转置卷积)、与编码器对应层级的特征图进行拼接(Skip Connection/跳跃连接)、以及两个3×3卷积 + ReLU 块组成。
- 跳跃连接(Skip Connections)是U-Net最关键、最具创新性的设计!其作用是将编码器阶段提取的、包含丰富空间细节的浅层特征图,直接传递到解码器中与之对称的层级并进行拼接。目的是解决信息丢失问题:下采样会导致位置细节信息丢失,跳跃连接将这些细节直接提供给上采样过程,帮助网络更精确地重建目标边界。解决缓解梯度消失问题:为梯度提供了更短的传播路径,使网络更易于训练。融合多尺度特征:将深层的抽象语义信息与浅层的细节外观信息相结合。
- 最终输出层为网络最后一层,使用一个1×1卷积,将64通道的特征图映射到K个输出通道(K代表分割的类别数)。对于二分类问题,通常使用Sigmoid激活函数;对于多分类问题,则使用Softmax。
3×3卷积是最经典、最常用的卷积操作。3×3卷积可直观理解为一个小窗口扫描器(我们称之为“卷积核”或“滤波器”)在一张大图片上从左到右、从上到下地滑动。每滑动到一个位置,就停下来,透过这个小玻璃片看下面覆盖的9个像素。其核心功能是提取空间特征,如边缘、角点、纹理等。多个3×3卷积层堆叠起来,可以逐步提取更复杂、更抽象的特征(例如,从边缘 -> 轮廓 -> 物体部件 -> 整个物体)。
对于一个1×1卷积,以一张RGB图像为例,其形状通常是:(高度 Height, 宽度 Width, 通道数 Channels=3)。输入1个像素点,但这个像素点不是一个单一的值,而是一个包含3个数字的向量(每个数字代表一种RGB的强度),值 = [R, G, B] (3个通道)。这个1×1卷积核本身也是一个有3个权重的向量 [W_r, W_g, W_b] 。它的操作是将这个像素点的3维向量 与 卷积核的 3维权重向量 进行点积:R * W_r + G * W_g + B * W_b。然后加上偏置,输出一个单一的数值。而1×1卷积的强大之处在于它能处理任意数量的通道,对应于不同的抽象特征(比如“边缘”、“纹理”、“猫胡子”、“狗耳朵”等)。这个过程可以理解为:这个1×1卷积核在学习如何“混合”这多种抽象特征,从而创造出一种新的、更高级的复合特征。进行简单比喻,1×1卷积就像一个坐在总控室的经理,它不关心单个像素点的空间细节(那是3×3卷积侦探的工作),而是负责整合来自不同情报源(通道-对应于你希望模型识别出的物体类别的数量)的信息,并决定哪些信息重要,需要加强或削弱,或者如何组合它们。例如在U-Net输出层使用1×1卷积和sigmoid激活函数对细胞膜图像进行二分类。白色像素表示细胞膜边界,黑色像素表示背景或其他细胞结构。
下图直观地展示了U-Net的经典架构的文字描述:
输入图像 (例如 572 x 572 ):
│
├── 编码器/Encoder (下采样路径/Downsampling)...
│ │
│ ... (特征图尺寸减小,通道数增加)
│
├── Bottleneck (最底层,特征图最小,通道数最多)
│
└── 解码器/Decoder (上采样路径/Upsampling)...
│
... (通过与Skip Connection拼接,特征图尺寸增大,逐步恢复细节)
│
输出图像为分割图 (例如 388 x 388 x K) K是通道数,直接对应于你希望模型识别出的物体类别的数量。
2、主要特点与优势
- 适用于小数据集:通过使用数据增强(如弹性变形)和高效的网络设计,即使在标注数据有限的医学领域也能表现优异。
- 精确的边界分割:得益于跳跃连接,模型在分割目标的边缘处非常精确,这对于医学诊断至关重要。
- 端到端训练:输入是图像,输出是分割图,整个网络可以一起进行训练优化。
- 简单而高效:结构对称、优雅,易于理解和实现。
二、membrane数据集
在U-Net图像分割的上下文中,membrane(膜/细胞膜 英 [ˈmembreɪn])通常指的是生物医学图像中的细胞膜分割任务。
1. 数据集结构
数据集data/membrane/ 目录通常包含:
data/membrane/
├── train/
│ ├── image/ # 原始训练图像
│ └── label/ # 对应的分割掩码(标签)
└── test 测试图像
└── test_outpu (预测结果会保存到这里)
2. 典型示例
输入图像为显微镜下的细胞群图像,输出标签为二值掩码,其中黑色像素表示细胞膜边界,白色像素表示背景或其他细胞结构。
以下图例为本案例的输入和输出:左图为输入、右图为输出。
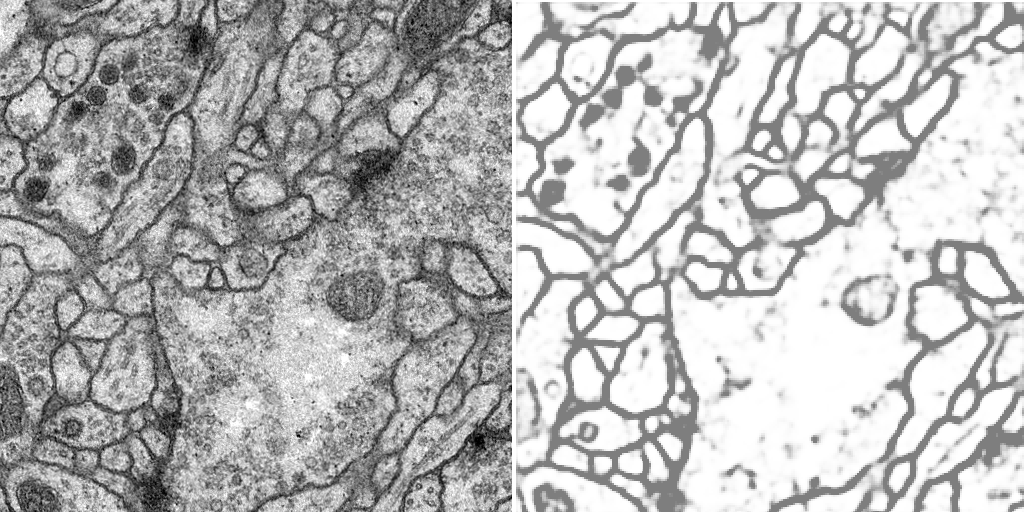
3. 数据集价值
基于该数据集,图像语义分割任务对于:细胞计数和形态分析、疾病诊断(如癌细胞检测)、药物研发(观察药物对细胞膜的影响)、生物学研究(细胞分裂、迁移等过程分析)都具有十分重要意义。
4. 经典数据集
著名的membrane分割数据集包括:
- ISBI 2012:电子显微镜图像中的神经元结构分割
- DSB2018:细胞核分割挑战赛数据
- 各种生物医学研究机构提供的细胞图像数据
三、工作流程
1、安装信息
通个命令行方式获取的开发包版本信息:
python -c "import PIL; print('PIL version:', PIL.__version__)"
PIL version: 8.3.1
python -c "import skimage; print('skimage version:', skimage.__version__)"
skimage version: 0.15.0
python -c "import tensorflow as tf; print('TF version:', tf.__version__)"
TF version: 1.14.0
2、模型定义
源码:model.py
a、U-Net编码器(下采样路径)
# 第一层:64个滤波器/卷积核
# 接受输入图像,用64个3x3的核进行卷积,并应用ReLU激活,输出一个与输入尺寸相同但深度为64的特征图。
conv1 = Conv2D(64, 3, activation='relu', padding='same')(inputs)
# 为什么要堆叠两个卷积层?
# 这是一种常见且强大的设计模式。两层3x3卷积串联的效果,近似于一层5x5卷积的感受野(即一个像素能看到原始输入中更大的区域),但拥有更少的参数和更强的非线性能力。
conv1 = Conv2D(64, 3, activation='relu', padding='same')(conv1)
# 为什么要进行下采样(池化)?
# 减小计算量: 特征图尺寸变小,后续层的计算负担大大减轻。
# 扩大感受野: 让后续层的一个像素能够“看到”前一层更大区域的信息。
# 引入平移不变性: 使网络对目标位置的微小变化不那么敏感,更关注特征的存在而非其精确位置。
pool1 = MaxPooling2D(pool_size=(2, 2))(conv1) # 下采样
# 第二层:128个滤波器/卷积核
conv2 = Conv2D(128, 3, activation='relu', padding='same')(pool1)
conv2 = Conv2D(128, 3, activation='relu', padding='same')(conv2)
pool2 = MaxPooling2D(pool_size=(2, 2))(conv2)
# 依此类推:256 → 512 → 1024
编码器特点:
- 每层滤波器数量翻倍:64→128→256→512→1024
- 使用最大池化进行2倍下采样
- 在深层添加Dropout防止过拟合
b、U-Net解码器(上采样路径)
# 上采样 + 跳跃连接
up6 = Conv2D(512, 2, activation='relu', padding='same')(
UpSampling2D(size=(2,2))(drop5)) # 上采样
merge6 = concatenate([drop4, up6], axis=3) # 跳跃连接
conv6 = Conv2D(512, 3, activation='relu', padding='same')(merge6)
conv6 = Conv2D(512, 3, activation='relu', padding='same')(conv6)
解码器特点:
- 每层滤波器数量减半:1024→512→256→128→64
- 使用上采样恢复空间分辨率
- 通过跳跃连接融合编码器的特征信息
c、输出层
conv9 = Conv2D(2, 3, activation='relu', padding='same')(conv9)
conv10 = Conv2D(1, 1, activation='sigmoid')(conv9) # 二分类输出
最终使用1×1卷积和sigmoid激活函数进行二分类。
d、模型编译
model.compile(optimizer=Adam(lr=1e-4),
loss='binary_crossentropy',
metrics=['accuracy'])
e、完整模型定义
U-Net模型架构定义如下:
def unet(pretrained_weights = None,input_size = (64,64,1)):
inputs = Input(input_size)
conv1 = Conv2D(64, 3, activation = 'relu', padding = 'same', kernel_initializer = 'he_normal')(inputs)
conv1 = Conv2D(64, 3, activation = 'relu', padding = 'same', kernel_initializer = 'he_normal')(conv1)
pool1 = MaxPooling2D(pool_size=(2, 2))(conv1)
conv2 = Conv2D(128, 3, activation = 'relu', padding = 'same', kernel_initializer = 'he_normal')(pool1)
conv2 = Conv2D(128, 3, activation = 'relu', padding = 'same', kernel_initializer = 'he_normal')(conv2)
pool2 = MaxPooling2D(pool_size=(2, 2))(conv2)
conv3 = Conv2D(256, 3, activation = 'relu', padding = 'same', kernel_initializer = 'he_normal')(pool2)
conv3 = Conv2D(256, 3, activation = 'relu', padding = 'same', kernel_initializer = 'he_normal')(conv3)
pool3 = MaxPooling2D(pool_size=(2, 2))(conv3)
conv4 = Conv2D(512, 3, activation = 'relu', padding = 'same', kernel_initializer = 'he_normal')(pool3)
conv4 = Conv2D(512, 3, activation = 'relu', padding = 'same', kernel_initializer = 'he_normal')(conv4)
drop4 = Dropout(0.5)(conv4)
pool4 = MaxPooling2D(pool_size=(2, 2))(drop4)
conv5 = Conv2D(1024, 3, activation = 'relu', padding = 'same', kernel_initializer = 'he_normal')(pool4)
conv5 = Conv2D(1024, 3, activation = 'relu', padding = 'same', kernel_initializer = 'he_normal')(conv5)
drop5 = Dropout(0.5)(conv5)
up6 = Conv2D(512, 2, activation = 'relu', padding = 'same', kernel_initializer = 'he_normal')(UpSampling2D(size = (2,2))(drop5))
merge6 = concatenate([drop4,up6], axis = 3)
conv6 = Conv2D(512, 3, activation = 'relu', padding = 'same', kernel_initializer = 'he_normal')(merge6)
conv6 = Conv2D(512, 3, activation = 'relu', padding = 'same', kernel_initializer = 'he_normal')(conv6)
up7 = Conv2D(256, 2, activation = 'relu', padding = 'same', kernel_initializer = 'he_normal')(UpSampling2D(size = (2,2))(conv6))
merge7 = concatenate([conv3,up7], axis = 3)
conv7 = Conv2D(256, 3, activation = 'relu', padding = 'same', kernel_initializer = 'he_normal')(merge7)
conv7 = Conv2D(256, 3, activation = 'relu', padding = 'same', kernel_initializer = 'he_normal')(conv7)
up8 = Conv2D(128, 2, activation = 'relu', padding = 'same', kernel_initializer = 'he_normal')(UpSampling2D(size = (2,2))(conv7))
merge8 = concatenate([conv2,up8], axis = 3)
conv8 = Conv2D(128, 3, activation = 'relu', padding = 'same', kernel_initializer = 'he_normal')(merge8)
conv8 = Conv2D(128, 3, activation = 'relu', padding = 'same', kernel_initializer = 'he_normal')(conv8)
up9 = Conv2D(64, 2, activation = 'relu', padding = 'same', kernel_initializer = 'he_normal')(UpSampling2D(size = (2,2))(conv8))
merge9 = concatenate([conv1,up9], axis = 3)
conv9 = Conv2D(64, 3, activation = 'relu', padding = 'same', kernel_initializer = 'he_normal')(merge9)
conv9 = Conv2D(64, 3, activation = 'relu', padding = 'same', kernel_initializer = 'he_normal')(conv9)
conv9 = Conv2D(2, 3, activation = 'relu', padding = 'same', kernel_initializer = 'he_normal')(conv9)
conv10 = Conv2D(1, 1, activation = 'sigmoid')(conv9)
model = Model(input = inputs, output = conv10)
model.compile(optimizer = Adam(lr = 1e-4), loss = 'binary_crossentropy', metrics = ['accuracy'])
#model.summary()
if(pretrained_weights):
model.load_weights(pretrained_weights)
return model
3、数据预处理模块
源码:data.py
a、颜色定义
Sky = [128,128,128]
Building = [128,0,0]
Pole = [192,192,128]
Road = [128,64,128]
Pavement = [60,40,222]
Tree = [128,128,0]
SignSymbol = [192,128,128]
Fence = [64,64,128]
Car = [64,0,128]
Pedestrian = [64,64,0]
Bicyclist = [0,128,192]
Unlabelled = [0,0,0]
COLOR_DICT = np.array([Sky, Building, Pole, Road, Pavement,
Tree, SignSymbol, Fence, Car, Pedestrian, Bicyclist, Unlabelled])
定义多类别分割的颜色映射(用于可视化)
b、adjustData() – 数据标准化函数
def adjustData(img,mask,flag_multi_class,num_class):
c、trainGenerator() – 训练数据生成器
def trainGenerator(batch_size,train_path,image_folder,mask_folder,aug_dict,...):
创建两个并行的数据生成器:
from keras.preprocessing.image import ImageDataGenerator
image_datagen = ImageDataGenerator(**aug_dict)
mask_datagen = ImageDataGenerator(**aug_dict)
使用相同seed确保同步:
image_generator = image_datagen.flow_from_directory(..., seed=seed)
mask_generator = mask_datagen.flow_from_directory(..., seed=seed)
相同的随机种子保证图像和掩码应用相同的增强变换。
数据配对和预处理:
train_generator = zip(image_generator, mask_generator)
for (img,mask) in train_generator:
img,mask = adjustData(img,mask,flag_multi_class,num_class)
yield (img,mask)
d、testGenerator() – 测试数据生成器
def testGenerator(test_path,num_image=30,target_size=(256,256),...):
- 逐个读取测试图像
- 归一化并调整尺寸
- 添加批次维度:
img = np.reshape(img,(1,)+img.shape)
e、其他辅助函数
geneTrainNpy(): 生成numpy格式的训练数据labelVisualize(): 将预测结果转换为彩色图像saveResult(): 保存预测结果
4、训练流程
源码:trainUnet.ipynb 调用 data.py 和 model.py的函数。
a、数据生成器配置
myGene = trainGenerator(1, 'data/membrane/train', 'image', 'label',
data_gen_args, target_size=(64, 64))
- batch_size=1(适应小显存)
- target_size=(64,64)(原512×512太大)
b、模型创建与训练
model = unet(input_size=(64, 64, 1))
model_checkpoint = ModelCheckpoint('unet_membrane.hdf5',
monitor='loss', save_best_only=True)
model.fit_generator(myGene, steps_per_epoch=300, epochs=5,
callbacks=[model_checkpoint])
c、模型测试
testGene = testGenerator("data/membrane/test")
model.load_weights("unet_membrane.hdf5")
results = model.predict_generator(testGene, 30, verbose=1)
saveResult("data/membrane/test_output", results)
5、二值分割原理
# 模型输出:每个像素的[0,1]概率值
# 后处理:阈值化为0或1
prediction = model.predict(image) # 输出概率图
binary_mask = (prediction > 0.5).astype(np.uint8) # 二值化
这个U-Net实现完整展示了深度学习图像分割的典型流程,从数据预处理、模型构建到训练预测的全过程。
运行结果
源码:trainUnet.ipynb
Found 30 images belonging to 1 classes.
Epoch 1/5
Found 30 images belonging to 1 classes.
300/300 [==============================] - 119s 398ms/step - loss: 0.4471 - accuracy: 0.7965
Epoch 00001: loss improved from inf to 0.44714, saving model to unet_membrane.hdf5
Epoch 2/5
300/300 [==============================] - 121s 405ms/step - loss: 0.3886 - accuracy: 0.8216
Epoch 00002: loss improved from 0.44714 to 0.38861, saving model to unet_membrane.hdf5
Epoch 3/5
300/300 [==============================] - 127s 423ms/step - loss: 0.3749 - accuracy: 0.8306
Epoch 00003: loss improved from 0.38861 to 0.37489, saving model to unet_membrane.hdf5
Epoch 4/5
300/300 [==============================] - 144s 480ms/step - loss: 0.3633 - accuracy: 0.8374
Epoch 00004: loss improved from 0.37489 to 0.36327, saving model to unet_membrane.hdf5
Epoch 5/5
300/300 [==============================] - 165s 550ms/step - loss: 0.3533 - accuracy: 0.8421
Epoch 00005: loss improved from 0.36327 to 0.35330, saving model to unet_membrane.hdf5
输入输出的图片样例,请参见第二章节中的典型示例部分。
源码开源协议
MIT License
Copyright (c) 2019 zhixuhao